最近由于临床报告中需要展示一些Uniprot的相关信息字段,因此需要对Uniprot中的部分重要信息进行获取,从而后续实现数据的本地化。因此需要对Uniprot数据库的使用方法和数据查询的机制进行了一些了解,记录如下
数据库简介
维护机构
通用蛋白质资源(UniProt)是蛋白质序列和注释数据的综合资源。 UniProt数据库包括UniProtKB (UniProt知识库)、UniRef (UniProt Reference Clusters)和UniProt Archive (UniParc)。 UniProt是欧洲生物信息学研究所(EMBL-EBI)、SIB瑞士生物信息学研究所(SIB Swiss Institute of Bioinformatics)和蛋白质信息资源研究所(PIR)的合作项目。UniProt联盟和主办机构EMBL-EBI、SIB和PIR都致力于长期保存UniProt数据库。
EMBL-EBI和SIB共同用于生成Swiss-Prot**和TrEMBL**, PIR生成Protein Sequence Database (PIR- psd)。 这两个数据集同时存在,但蛋白质序列覆盖范围和注释优先级不同。 TrEMBL(翻译后的EMBL核苷酸序列数据库)最初创建是因为序列数据的生成速度超过了Swiss-Prot的能力。 与此同时,PIR维护了PIR- psd及相关数据库,包括蛋白质序列数据库iProClass和整理的家族库。 2002年,这三家机构决定集中他们的资源和专业知识,成立了UniProt联盟。
- EBI( European Bioinformatics Institute):欧洲生物信息学研究所(EMBL-EBI)是欧洲生命科学旗舰实验室EMBL的一部分。位于英国剑桥欣克斯顿的惠康基因组校园内,是世界上基因组学领域最强地带之一。
- SIB(the Swiss Institute of Bioinformatics):瑞士日内瓦的SIB维护着ExPASy(专家蛋白质分析系统)服务器,这里包含有蛋白质组学工具和数据库的主要资源。
- PIR(Protein Information Resource):PIR由美国国家生物医学研究基金会(NBRF)于1984年成立,旨在协助研究人员识别和解释蛋白质序列信息。
数据库组成
截至目前数据库共包含4个subDatabase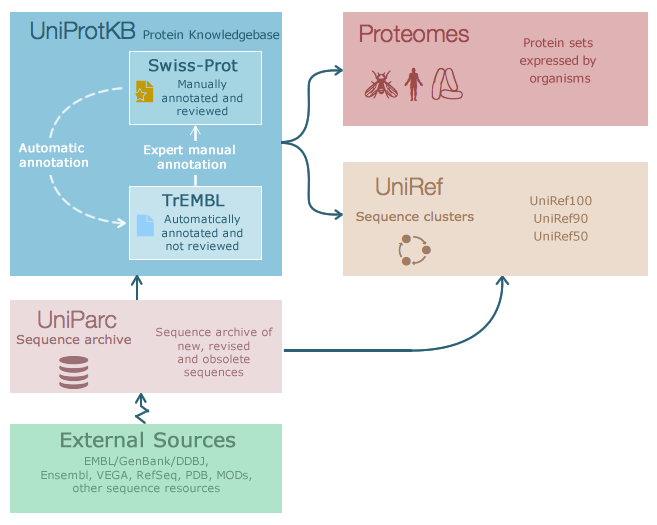
- The UniProt Knowledgebase (UniProtKB)
UniProt知识库, 特别是UniProtKB/Swiss-Prot,被用来访问蛋白质的功能信息。 每个UniProtKB条目都包含了氨基酸序列、蛋白质名称或描述、分类数据和引文信息,除此之外,我们还添加了尽可能多的注释。 这包括广泛接受的生物本体、分类和交叉引用,以及以实验和计算数据的证据归因的形式明确标注标注质量。 UniProt Reference Clusters (UniRef)
UniRef数据库提供来自UniProtKB和UniParc记录的聚类序列集,以提供多个分辨率的序列空间的完整覆盖。 UniRef90和UniRef50的数据库大小分别减少了约40%和65%,提供了显著更快的序列搜索。UniProt Archive (UniParc)
UniParc是最全面的公开可访问的非冗余蛋白质序列数据库,提供这些序列的所有潜在来源和版本的链接。 你可以立即发现一个感兴趣的序列是否已经在公共领域,如果不是,就找出它最近的亲属。UniProt Metagenomic and Environmental Sequences (UniMES)
UniMES是一个专门存储宏基因组和环境数据的数据库。
更多详细信息可以参考官方说明文档
数据库使用
UniProtKB 的使用
由于本次数据的获取信息,均来自于Uniport的知识库,目前主要针对知识库进行介绍。
进入知识库的主页,看到的信息如下图: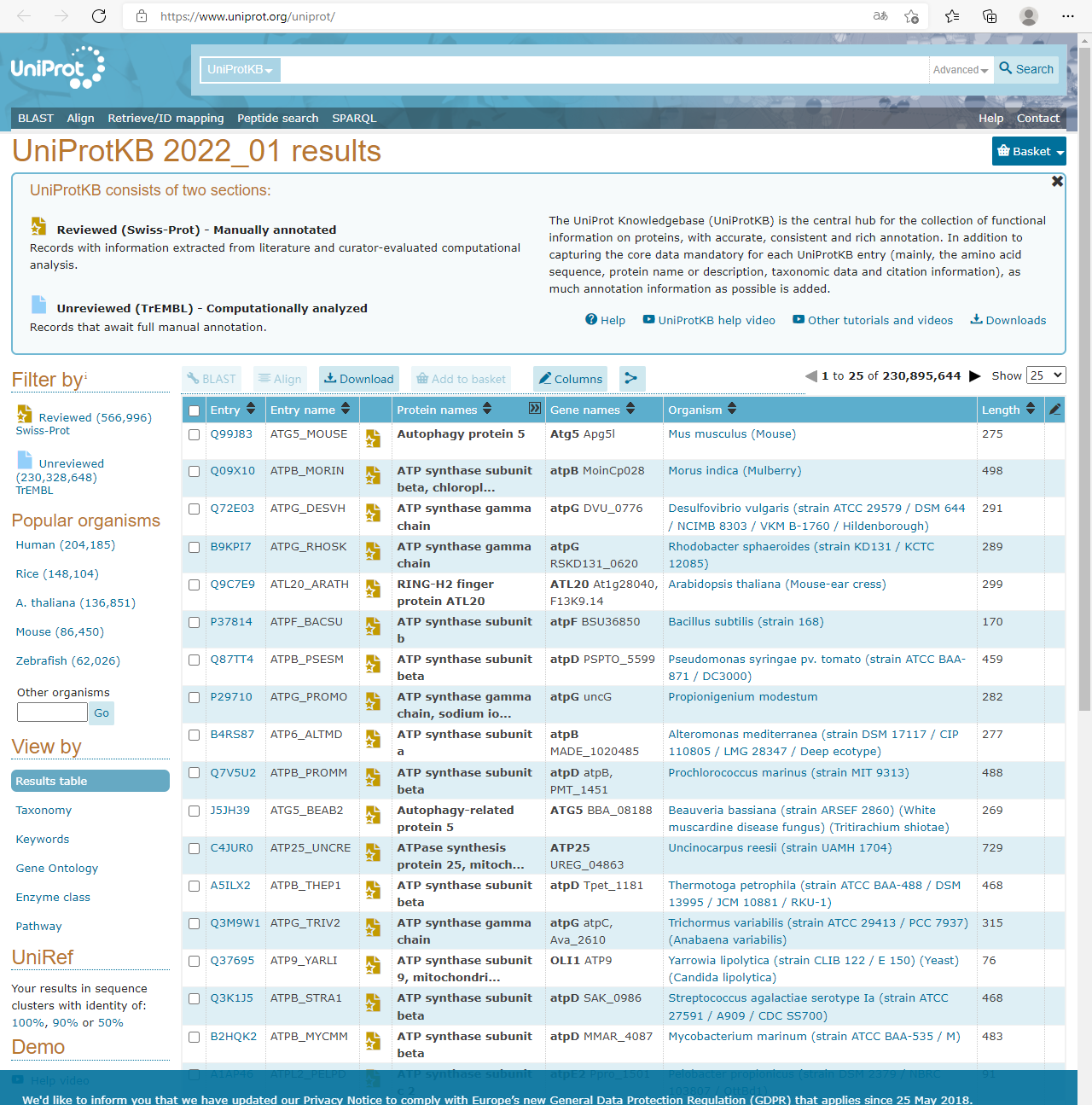
- 最上面是 搜索框,
- 左侧可以进行数据的过滤,例如肿瘤数据关注点主要是人的基因信息,可以直接选择Human剔除掉一些非人源的数据信息。
- 中间就是整个数据库数据的展示了。
重点介绍数据内容主题框的上面两个功能 Download 和 Columns
- Download
字面意思,进行数据的下载,可以选择多种数据格式进行数据下载,tsv、gff、xml、fasta等等,我们可以根据需要选择相关格式进行下载 - Columns
这个功能可以说是一个非常人性化的功能,尤其是结合Download,可以完全不适用爬虫获取该数据库的所有需要的信息,点击进入Columns后,可以筛选在汇总表格中需要展示的字段信息(具体那些字段需要,可以在详细表中进行获取,毕竟下数据了,我们首先要知道获取什么数据)。
示例如下图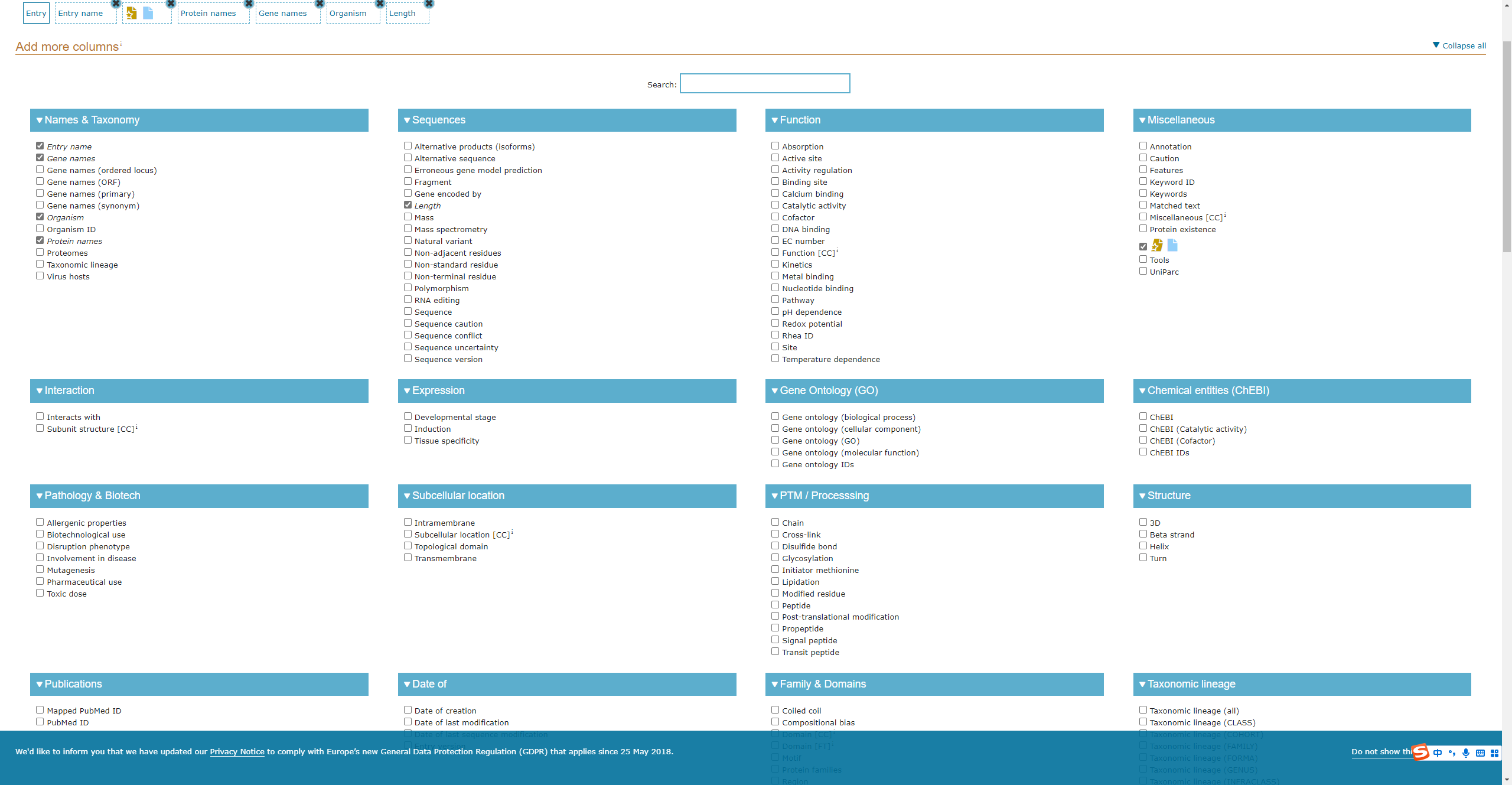
勾选我们需要的信息后,点击下方的 save, 就可以在内容中现实特定的信息,结合Download,可以实现快速的数据获取。
获取信息后,在进行简单的格式整理,就可以直接使用了,相比那些验收、IP检测、流量限制等方案层出不确定网站,可以说是非常友好了。
BGI的下载与处理
需要的信息列:
- Entry (Names & Taxonomy)
- Entry name (Names & Taxonomy)
- Gene names (Names & Taxonomy)
- Organism (Names & Taxonomy)
- Length (Sequences)
- Repeat (Family & Domains)
- Region (Family & Domains)
- Zinc finger (Family & Domains)
- Domain [FT] (Family & Domains)
- Nucleotide binding (Function)
- Cross-reference (GeneID)
- DNA binding (Function)
基于肿瘤2022.3.1的解读需求,可以参考进行下载%20[9606]%22&format=tab&force=true&columns=id,entry%20name,genes,organism,length,feature(REPEAT),feature(REGION),feature(ZINC%20FINGER),feature(DOMAIN%20EXTENT),feature(NP%20BIND),database(GeneID)&sort=score&compress=yes)
下载后,流程处理脚本使用 toolkits/07.DealWithDatabase/UniprotKB_DataClean.py(GitHub仓库) 对数据进行处理。
处理后的文件结果示例如下:
| Gene | GeneID | GeneLength | feature_key | Position_region | Uniport_description |
|---|---|---|---|---|---|
| BLK | 640; | 505 | Region | 1..37; | “Disordered” |
| BLK | 640; | 505 | Domain [FT] | 58..118; | “SH3” |
| BLK | 640; | 505 | Domain [FT] | 124..220; | “SH2” |
| BLK | 640; | 505 | Domain [FT] | 241..494; | “Protein kinase” |
| BLK | 640; | 505 | Nucleotide binding | 247..255; | “ATP” |
| BDP1 KIAA1241 KIAA1689 TFNR | 55814; | 2624 | Repeat | 823..877; | “1; approximate” |
| BDP1 KIAA1241 KIAA1689 TFNR | 55814; | 2624 | Repeat | 878..932; | “2” |
| BDP1 KIAA1241 KIAA1689 TFNR | 55814; | 2624 | Repeat | 933..987; | “3” |
| BDP1 KIAA1241 KIAA1689 TFNR | 55814; | 2624 | Repeat | 988..1040; | “4” |
| BDP1 KIAA1241 KIAA1689 TFNR | 55814; | 2624 | Repeat | 1041..1094; | “5” |
| BDP1 KIAA1241 KIAA1689 TFNR | 55814; | 2624 | Repeat | 1095..1148; | “6” |
| BDP1 KIAA1241 KIAA1689 TFNR | 55814; | 2624 | Repeat | 1149..1203; | “7” |
| BDP1 KIAA1241 KIAA1689 TFNR | 55814; | 2624 | Repeat | 1204..1257; | “8; approximate” |
| BDP1 KIAA1241 KIAA1689 TFNR | 55814; | 2624 | Repeat | 1258..1327; | “9; approximate” |
| BDP1 KIAA1241 KIAA1689 TFNR | 55814; | 2624 | Region | 1..299; | “Interaction with ZBTB43” |
| BDP1 KIAA1241 KIAA1689 TFNR | 55814; | 2624 | Region | 1..142; | “Disordered” |
| BDP1 KIAA1241 KIAA1689 TFNR | 55814; | 2624 | Region | 193..241; | “Disordered” |
| BDP1 KIAA1241 KIAA1689 TFNR | 55814; | 2624 | Region | 355..470; | “Required for phosphorylation by CSNK2A1” |
| BDP1 KIAA1241 KIAA1689 TFNR | 55814; | 2624 | Region | 379..449; | “Disordered” |