之前也一直在使用阿里云的基因分析平台,但是更多是作为解决方案的技术储备,而且其实更多的是平台迁移,数据都比较常规。而最近接手到一些样本量非常大的非常规项目(单样本有接近2T的数据),这个数据量,不管是计算资源还是存储资源,其实本地IDC都不太能支持,所以将视角转向通过基因分析平台进行实现。由于数据规模超大,也因此遇到许多新的问题,所以趁此机会介绍一下基因分析平台的使用方法和一些隐藏的坑。
基因分析平台简介
适用场景
只要涉及数据,没有合规性限制,上云可能都整体是一个更稳定更省心的解决方案。
首先自建机房,往往在资源上需要进行过饱和的投入,以应对业务峰值和未来的业务成长场景,而这也就带来了低谷期的业务浪费。简单说,要么一直有资源浪费,要么经常资源不足。这也是本地IDC搭建在底层逻辑上无法解决的问题。 而且自建机房,除了硬件资源外,还需要机房(静电地板、空调、备用发电机、安保、网络、电力)、各类运维管理人员投入、设备的阶段性维护投入等。对于小型企业来讲,自建集群在前期需要一笔不菲的投入。
针对业务量稳定的企业,使用本地自建IDC集群,对集群有更高的可控性,资源可以稳定在饱和运行的水平,本地IDC的性价比会比较高。但是现实中这类情况非常少,甚至一些稳定业务也是周期性稳定,业务低谷无法避免会存在资源的浪费。
所以其实现在针对大型企业(有资源使用量的基本盘,但是依然存在顶峰波动的情况)混合云是一个比较不错的选择,业务执行逻辑优先充分利用本地资源,资源不足时,进行云商资源的弹性扩展应对峰值需求。
使用方式
基因分析平台就是阿里云针对基因检测领域开发的云计算平台,面向基因检测的垂直细分领域进行了一些适配,如果大家本地使用WDL和docker的话,可以实现分析流程的无缝迁移。基因分析平台工作模式如下: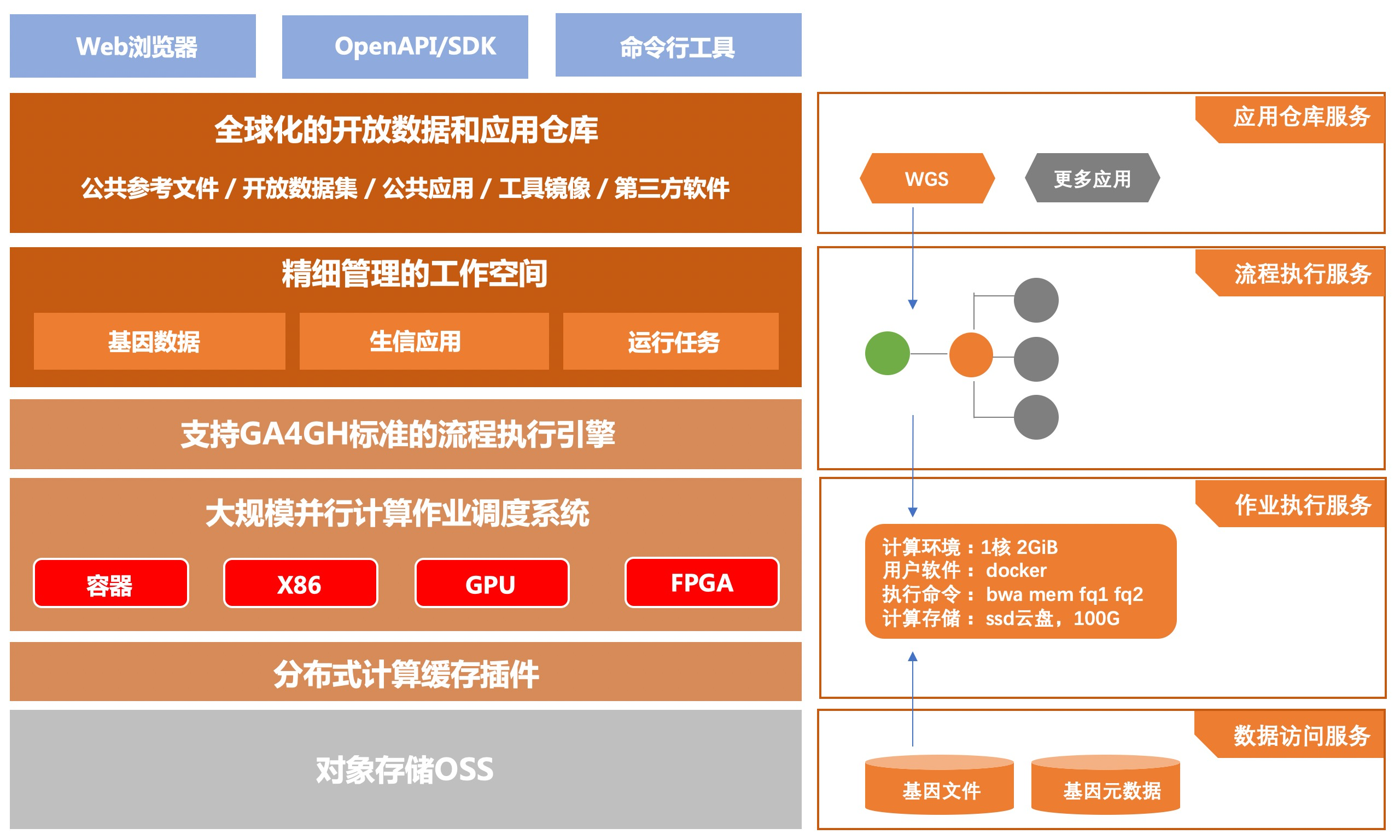
基因分析平台本身采用的模式是,存算分离,按需分配资源的模式。每个任务的计算资源、存储资源、执行环境都是作为独立的资源项,进行单独的配置。每个资源项支持的上限(64核心 512G内存,512T存储)还是蛮高的。然后再任务执行阶段,想一块块积木进行组装,然后进行相关的分析。
基因分析平台,是工作空间作为一个整体管理一个独立完整的项目,以下是工作空间主要包含的几个内容,如果大家第一次接触,主要关注文件、应用、运行即可,另外的表格、模板、投递三部分属于批量任务投递的部分,可以放到后面进行了解,
| 类别 | 名称 | 用途 | 含义 |
|-|-|-|-|
| 数据管理 | 文件 | OSS存储 | 账号可以访问的OSS存储,可以直接查看OSS的文件 |
| 应用配置 | 应用 | 分析流程 | 记录了流程的wdl文件和对应的说明文档、版本信息等 |
| 分析任务 | 运行 | 流程执行 | 启动一个分析流程,执行分析任务,查看任务进度和状态 |
| 数据管理 | 表格 | 表格文件 | 用户按特定表格模板上传的表格文件,用于批量进行任务投递 |
| 应用配置 | 模板 | 投递模板 | 基于应用和表格文件,配置好的任务投递模板,可以直接用于任务投递 |
| 分析任务 | 投递 | 流程投递 | 基于应用和表格,启动一批分析任务,执行分析 |
因此如果我们想使用基因分析平台进行任务分析,就需要做一些准备工作:
- 需要将我们要分析的数据存储到OSS上,所有输入输出数据的长期保存都依赖于OSS,分析中挂载的存储上产生的中间数据均不可见。
- 搭建一个分析流程,分析流程目前仅支持 WDL ,调用的分析环境(docker镜像或阿里提供的software)、计算资源(最小 1c2Gb 到最大 64c512Gb )、分析存储(HDD和SSD)都是在WDL中指定的。
性能监控
云平台
注意事项
流程部署过程中,WDL语法中所有的File类型会作为文件关联到分析容器内(/cromwell_inputs/ 目录下),所以一定要明确数据的真实类型,错误的类型使用,会导致分析过程中出现文件缺失。
由于流程运行环境依赖于容器,因此需要注意相关的容器需要开放访问权限。
wdl本身提供了scatter语法,以便进行一些遍历/并行操作,但是在基因分析平台任务监控中,只能进行2层深度的scatter解析(多深度可以进行任务的分析,但是并没有对应颗粒度的任务状态监控),所以为了更好的查看所有任务的运行情况和资源使用情况,最好控制wdl的数据深度。比如最高维度可以借助投递逻辑来实现(比如下述示例中的Sample_info层),流程只是针对一个样本,批次多样本的投递依赖于表格和模板结合进行投递的方式实现。
1
2
3
4
5
6
7scatter (Sample_info in Samples_info){
scatter (sample_lib in Sample_info){
scatter (Fastq_info in sample_lib.fastqlist){
call xxxx ;
}
}
}
历史的一些隐藏小坑
基因分析平台对 wdl 文件大小有一个非明文限制(100kb以下),23年2月初和阿里反馈调整过一次,目前我们没有出现由于文件大小导致的问题,但是限制可能依然存在。不过常规流程可能不会遇到文件大小瓶颈问题。
系统盘(local-disk)的存储范围 40G~500G,超出后无法启动任务,可以不配置(默认40G),如果为了规避资源浪费设置了10G资源,会导致任务无法启动。
数据盘官网提示可以挂载 16*32T,但是自定义挂载目录(例如说明文档提供的 /mnt1 、/mnt2),那么生成结果无法正常导入OSS,数据盘挂载目录只有使用 /cromwell_root 时且output中文件名不指定根目录时,生成的数据才能正常导出OSS,并被作为正常输入被下游任务调用。 如果使用根目录,会导出到OSS,但是文件路径会有问题影响下游调用。为方便理解,提供一个示例供大家参考。
1 | task test_run { |