NGS在数据下机前需要对数据进行一些列的实验处理后才能进行上机测序,因此了解实验原理才能更好的从根源了解数据处理过程,及各个环节的不同处理方案可能带来的优劣影响。在下游数据处理过程中,更有针对性的对数据处理过程进行优化提升。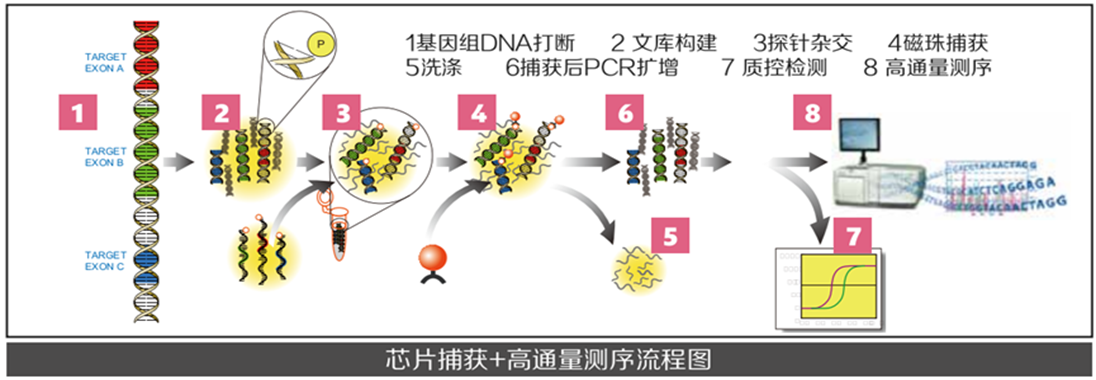
DNA的提取
细胞裂解
预处理
加入保护液(如TE buffer)来溶解DNA并防止其降解;在破胞前加入去垢剂来去除杂质并破坏细胞(如SDS是一种表面活性剂,能破坏细胞膜上的脂质,并在低温下使其沉淀)
主要方法
机械破碎法(振荡珠磨、液氮研磨、反复冻融等)和酶解法(如溶菌酶溶解细菌细胞壁)
去除杂质
杂质
如细胞碎片、蛋白质、RNA、腐殖酸等
方法
化学法(加入抑制因子沉淀杂质、使用氯仿等有机溶剂溶解杂质)、酶解法(例如蛋白酶K、RNA酶降解蛋白质、RNA)
回收DNA
主要方法
- 醇沉淀法:DNA不溶解于异丙醇、乙醇等
- 过柱收集法:DNA在高盐环境下可以吸附在硅胶滤膜上,这是大部分试剂盒采用的方法
- 磁珠吸附法:DNA分子通过氢键吸附到具有磁性的磁珠上,然后在磁场中分离磁珠,常见于自动提取仪
清洗溶解DNA
方法
- 对沉淀的DNA、收集柱DNA以及磁珠吸附的DNA使用70%乙醇清洗;
- 待乙醇挥发后使用无菌水溶解DNA。
DNA提取原则
- 保证核酸一级结构的完整性;
- 核酸样品中不应存在对酶有抑制作用的有机溶剂和过高浓度的金属离子;
- 其他生物大分子如蛋白质、多糖和脂类分子的污染应降低到最低程度;
- 其他核酸分子,如RNA,也应尽量去除。
文库制备
末端修饰
- 使用Taq聚合酶补齐不平的末端;
- 并在两个末端添加突出的碱基A,从而产生粘性末端(若使用Taq酶扩增,则无需末端修饰);
- 产生粘性末端的片段可以添加接头(Adaptor)。
添加接头
- 经过末端修饰后的PCR片段末端具有突出的A尾,而接头具有突出的T尾,可以使用连接酶将接头添加到DNA片段两端。
- NEB的接头为特殊的碱基U连接的环状结构(可以增强稳定性),因此连接接头后,还需要将碱基U删除从而形成“Y”形接头。
- 上一步添加的接头主要是为了后续PCR中作为引物扩增继续添加文库index和与测序平台互补的寡核苷酸序列(此外还作为测序引物Rd1 SP/Rd2 SP)。
- 之所以为“Y”型开叉结构,是因为每一端接头是两条不互补的序列(每一端都是Rd1 SP与Rd2 SP交错),连接酶没有选择性,每个接头都是只靠突出的T来与DNA连接,“Y”接头保证了每条单序列两端均为不同的测序引物,从而在后续PCR中可以连接不同的寡核苷酸序列(P5/P7)。
过程示意图如下: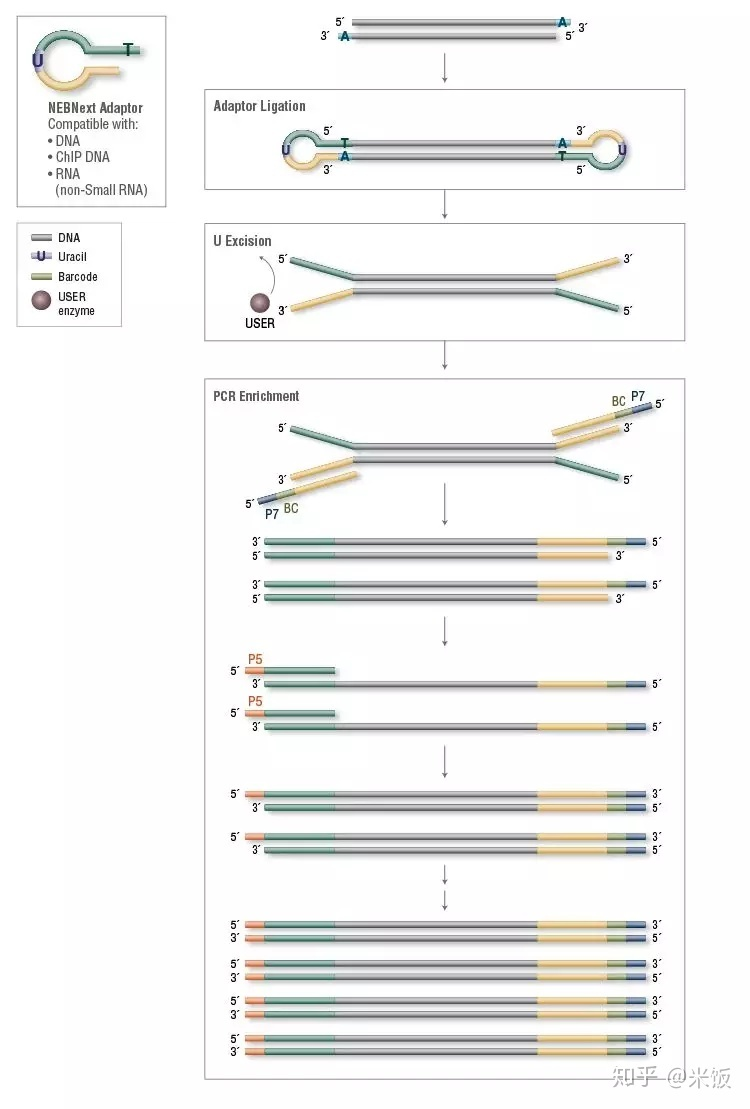
磁珠纯化
目的
添加接头后的文库体系中含有聚合酶、连接酶等各种酶以及辅助物质,接头的添加也是过量的,而且由于末端的不稳定性,容易形成自连片段,鸟枪法打断的片段中也可能有大片段存在,所以需要特殊磁珠(AMPure XP Beads)纯化来去除大片段以及各种杂质,从而获得成功添加接头的文库片段。
原理
磁珠可以通过氢键等作用力来吸附DNA片段,磁珠本身不具有片段大小选择的能力,但其储存的buffer里面含有20%的PEG 8000,PEG浓度越大则可以吸附的DNA片段越小。
注意事项
磁珠纯化的时候要根据文库片段不同严格控制磁珠添加量(其实是PEG添加量)来实现片段选择。
PCR扩增
- 添加了接头的DNA片段,可以使用与接头互补的引物来扩增。
- 此外,片段还需要添加用于区分不同文库的特异性index,以及与测序仪芯片互补的两种寡核苷酸序列(P5/P7)。
第二次磁珠纯化
- PCR后需要将产物DNA片段与聚合酶等杂质分离,因此再次进行磁珠纯化。
- 之后进行质量检测,包括DNA浓度检测、琼脂糖凝胶
电泳和片段长度检测,完成建库。
NGS测序仪上机
待补充
参考来源
- https://mp.weixin.qq.com/s/zNFvod8B-VhX7Kq7OgoRMA
- ClarkeA C, Prost S, Stanton J a L, et al. From cheek swabs to consensus sequences: anA to Z protocol for high-throughput DNA sequencing of complete humanmitochondrial genomes[J]. Bmc Genomics, 2014, 15(1): 1-12.
- BowmanS K, Simon M D, Deaton A M, et al. Multiplexed Illumina sequencing librariesfrom picogram quantities of DNA[J]. Bmc Genomics, 2013, 14(1): 135-143.
- MardisE R. Next-Generation DNA Sequencing Methods[J]. Annual Review of Genomics &Human Genetics, 2008, 9(9): 387-402