什么是假基因
假基因是与功能基因具有显着同源性的 DNA 序列,但它们缺乏用于转录的启动子序列或包含其他阻止功能产物形成的突变,不能产生功能性蛋白的序列[1]。同时由于假基因通常不面临选择压力,因此更容易出现变异信号的积累。
根据 GENCODE统计数据,人类基因组中有14737个假基因(编码基因19393个),对应3391个 “parent gene”。当然其实截止目前,人类基因组的基因总数目其实也尚未在业界达成一个统一性的共识。但是通过GENCODE的数据,至少我们可以看到,在基因组中,假基因的数目占比非常巨大。
假基因的起源
提到假基因的产生,其实就绕不过新基因的产生(假基因只是一类不能产生功能性蛋白的特殊新基因)。而新基因的产生过程会涉及到各种分子事件,同时这些事件必须发生在种系中才能遗传给下一代。当新产生的基因能进行遗传后,变回参与到和环境的互作(压力选择)中,最终取得优势得以保存,或者存在劣势被淘汰。
假基因最初被定义为类似于已知基因但不能产生功能性蛋白质的序列,对假基因的研究不仅揭示了基因退化的频率,而且还揭示了许多曾经被认为是退化蛋白质编码基因的序列实际上是功能性 RNA 基因。
多年来,科学家提出了几种产生新基因的机制。这些包括基因复制、转座子蛋白驯化、横向基因转移、基因融合、基因裂变和从头起源[4]。
- Gene Duplication 基因复制
- Transposable Element Protein Domestication 转座子蛋白驯化
- Lateral Gene Transfer 横向基因转移
- Gene Fusion and Fission 基因融合与裂变
- De Novo Gene Origination 从头基因起源
假基因起源于与蛋白质编码基因相同的机制,随后是随后破坏阅读框或导致过早终止密码子插入的致残突变(例如,核苷酸插入、缺失和/或替代)的积累[2]。假基因可大致分为两类:
- 未加工的假基因通常包含内含子,并且它们通常位于其旁系同源基因的旁边。
- 加工过的假基因被认为起源于逆转录转座;因此,它们缺少内含子和启动子区域,但它们通常包含聚腺苷酸化信号,并且两侧是同向重复序列。逆转录错误和缺乏适当的监管环境通常会导致基因转录的退化。
给定基因组中假基因的丰度通常取决于基因复制和丢失的速率。哺乳动物似乎有大量经过处理的假基因——大约 8,000 个[3]。
假基因对分析带来的影响
基于假基因的产生机制,我们不难发现,假基因,尤其是通过基因复制、横向基因转移、基因融合等原因产生的假基因会和基因组上其他区域(假基因产生的母本)存在序列的高度同源性。而这些高度同源性的序列存在会使得NGS分析过程变得复杂化:
- 使用短读长(75-300 bp ),则序列同源性会导致假基因和母本片段区域难以进行区分。
序列同源使准确的reads比对(映射)变得复杂,如下图所示。映射到多个基因组位置的序列读数在分析中被丢弃,这会导致序列覆盖率出现缺口。
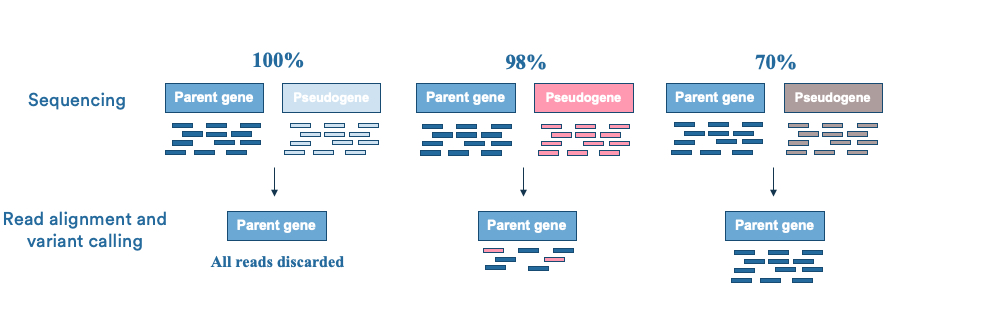
差异加大时,可以进行有效的区分,但是随着序列同源性增加时,会出现假基因的错误比对(假基因序列变动会被错误识别为变异)。当序列读数与几个基因组位置对齐得同样好时,它们将被丢弃。而在NGS的靶向捕获中,假基因的存在还会影响杂交捕获过程。如前所述,人类有上万个假基因(GENCODE 项目),如果我们的检测范围中存在假基因或者假基因对应的编码基因/同源区域,则会影响对应区域的检测准确性,这类区域的准确性会低于没有假基因影响的区域。
有什么方式可以减少假基因的影响
- 最简单直接的,是在进行芯片涉及阶段,尽可能剔除掉假基因和对应的同源区域及编码基因。从根本上消除假基因的干扰。想获取具体的假基因清单,可以在Ensemble中下载基因结构文件gff,其中基因的biotype 标签会记录基因是否属于假基因。
- 尽可能采用长读长测序(PE250>PE100>PE50),长度长测序比短读长能更有效的区分进行同源区域的识别和划分[5]。
- 提高比对质量值的阈值,仅保留高比对质量的数据进行变异的检出。
- 涉及长Long-Range PCR和Sanger 引物进行验证,确定分析结果。
- 进行生物信息流程的定制化,以消除影响。
Reference
[1]. Wilde CD. Pseudogenes. CRC Crit Rev Biochem. 1986;19(4):323-352.
[2]. D’Errico I, Gadaleta G, Saccone C. Pseudogenes in metazoa: origin and features. Brief Funct Genomic Proteomic. 2004;3(2):157-167. doi:10.1093/bfgp/3.2.157
[3]. Zhang Z, Carriero N, Gerstein M. Comparative analysis of processed pseudogenes in the mouse and human genomes. Trends Genet. 2004;20(2):62-67. doi:10.1016/j.tig.2003.12.005
[4]. Chitra Chandrasekaran, Esther Betrán .Origins of New Genes and Pseudogenes
[5]. Vahid Bahrambeigi and others, An Approach for Accurate Molecular Diagnosis of Highly Homologous SDHA Gene, American Journal of Clinical Pathology, Volume 146, Issue suppl_1, September 2016, 214
[6]. ensembl