参考文献
工作组
- ACMG、
- 分子病理协会(the Association for Molecular Pathology, AMP)
- 美国病理学家协会(the College of American Pathologists, CAP)
应用范围
基因分型、单基因、基因包、外显子组和基因组.
使用特定标准属于来描述孟德尔疾病相关的基因变异:
| ACMG Standards and Guidelines | 遗传变异分类标准与指南 |
|---|---|
| pathogenic | 致病的 |
| likely pathogenic | 可能致病的 |
| uncertain significance | 意义不明确的 |
| likely benign | 可能良性的 |
| benign | 良性的 |
建议
临床分子基因检测应在符合临床实验室改进修正案(CLIA)认证的实验室中进行, 其检测结果应由通过职业认证的临床分子遗传学家或分子遗传病理学家或相同职能的专业人员解读.
术语
突变: 是指核苷酸序列的永久性改变
多态性:指频率超过1%的变异。
命名
标准的基因变异命名由人类基因组变异协会(the Human Genome Variation Society,HGVS)维护和版本化(https://www.hgvs.org/mutnomen), 除非另有说明, 一般推荐该命名法作为确定变异命名的首要准。
当描述变异时,可利用这些工具提供正确的HGVS命名(http://mutalyzer.nl)。
编码命名应该使用翻译起始密码子ATG中的A作为位置编号1来描述。
协会支持的参考转录本通常可以通过LRG数据库(http://www.lrg-sequence.org)、CDS 共识数据库(https://www.ncbi.nlm.nih.gov/CCDS/CcdsBrowse.cgi) 、人类基因突变数据库(http://www.hgmd.cf.ac.uk) 、ClinVar(http://www.ncbi.nlm.nih.gov/clinvar)或特异基因座数据库来确定
文献及数据库使用
在使用人群数据库时,须明确数据库收录的是健康群体的信息还是患病群体的信息; (如能确认)数据库是否收录了同一家庭多名成员的信息以及数据库收录的受试者的年龄范围.
当使用数据库时, 临床实验室应做到:
- 确定数据库的更新频率, 确定数据库收录相关数据时是否进行了校勘, 以及采用什么方法进行数据校勘;
- 确认采用HGVS 命名体系, 并确定述变异的基因组版本和转录本参考序列;
- 确定数据分析准确度的验证程度(如变异是源自于低覆盖的新一代测序,还是通过了Sanger 测序验证), 并分析用于评估数据准确度的各种指标, 要获得这些信息可能需要阅读相关的文献;
- 确定收录对象的来源及其唯一性.
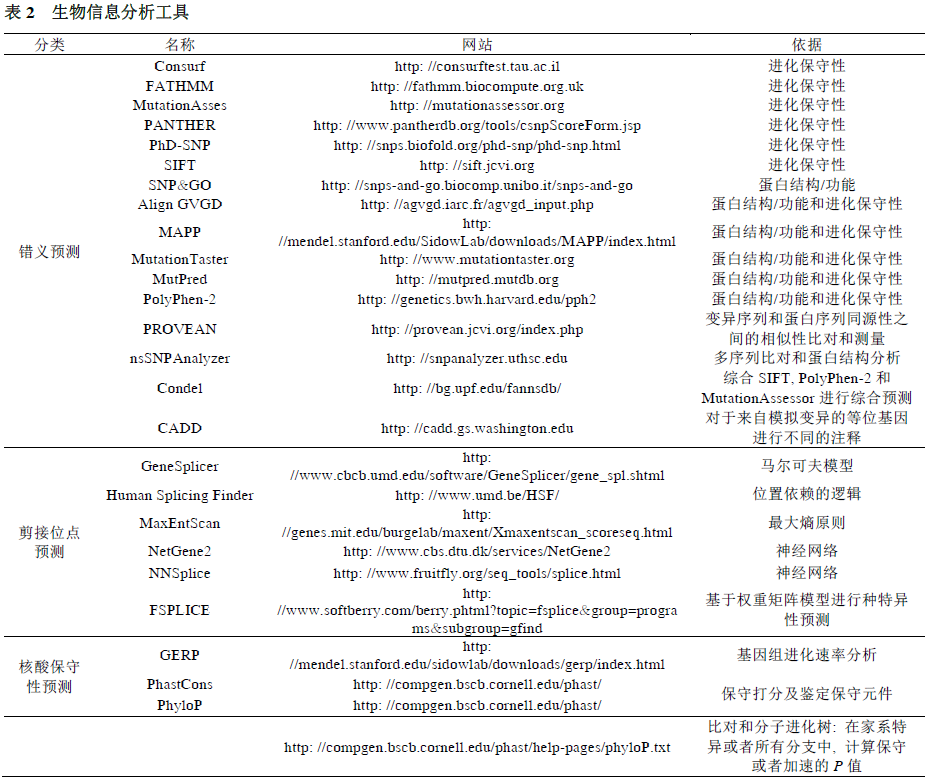
生物信息学计算预测程序
各种公共和商业化计算机工具可以辅助解读序列变异. 每种工具使用的算法可能有差异, 但都会包含序列变异在核苷酸及氨基酸水平上作用影响的判断, 包括变异对主要转录本, 可变转录本, 其他基因组元件影响作用的确认.
主要分为两类:
- 一类可以预测错义变异是否会破坏蛋白质的功能或结构;
- 另一种可以预测是否影响剪接
多数算法预测已知致病的错义突变的准确率能达65%~80%[12]. 但是大多数工具的特异性较低, 导致有些错义改变被过度预测为有害突变, 而且对于影响较小的错义变异的预测也不可靠[14]. 目前临床实验室常用的错义变异解读工具有PolyPhen 2[15],SIFT[16]和MutationTaster[17].
序列变异解读的拟定标准
以下评估变异证据的方法是用了解释在临床诊断实验室中具有疑似遗传(主要指孟德尔遗传)疾病患者的变异. 并不适用于解读体细胞变异、药物基因组(PGx)变异、或者是多基因非孟德尔复杂疾病相关的基因变异. 在外显子组或基因组研究中, 对候选基因(意义不明确的基因(GUS))应用这些准则时应当谨慎(见下面注意事项), 因为本指南目的不是满足鉴定新致病基因的研究需求.
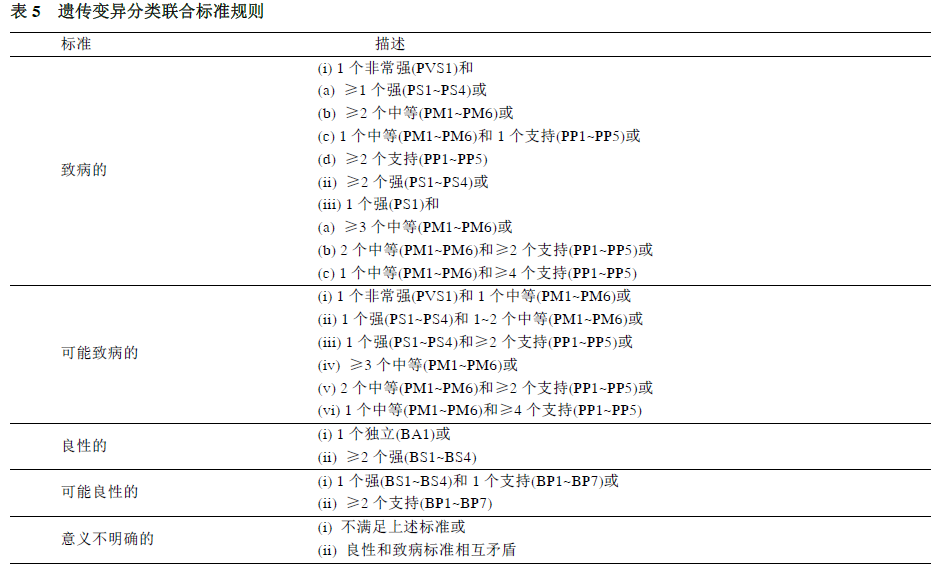
本指南提供了两套标准: 一是用于对致病或可能致病的变异进行分类(表3), 另一是用于对良性或可能良性的变异进行分类(表4). 致病变异标准可分为非常强(very strong, PVS1), 强(strong, PS1~4); 中等(moderate, PM1~6), 或辅助证据(supporting,PP1~5). 良性变异证据可分为独立(stand-alone, BA1),强(strong, BS1~4),和支持证据(BP1-7)
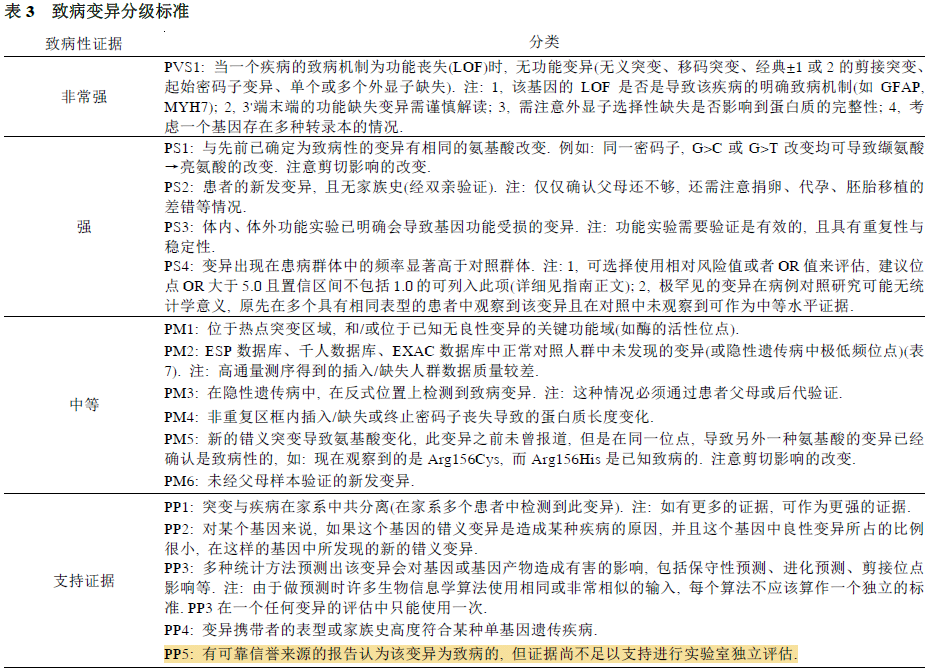

PVS1 极强致病性变异
从业人员需谨慎考虑以下原则:
- 当将该类变异归类为致病性时, 需确认无功能变异(null variants)是已知的致病机理, 且与该疾病的遗传模式相一致. 例如, 有些基因(如许多肥厚性心肌病基因)只有杂合错义突变时才致病, 而杂合无功能变异却是良性的. 仅基于这一项证据来看, 对显性肥厚性心肌病来说, MYH7 基因上出现一个新的杂合无义突变不一定是致病的, 而CFTR 基因上出现一个新的杂合无义突变则有可能是一个隐性致病变异.
- 当文献中将3′远端下游截短变异注释成致病突变时, 要特别小心. 特别是当所预测的终止密码子出现在最后一个外显子, 或者出现在倒数第二个外显子的最后50 个碱基对时, 这种无义突变介导的转录降解[22]可能不会发生, 这个蛋白很可能会表达.据此所预测的截短蛋白的长度也是致病性评估的因素, 但这些变异未经功能分析是无法进行判定的.
- 就剪接位点变异而言, 因外显子剪切位点的供体/受体位点改变或产生了新的剪切位点, 从而可能导致外显子丢失、缩短, 也可能会使内含子序列变成外显子部分. 虽然剪切位点变异可能被预测为无功能变异, 然而该变异类型造成的影响需要通过RNA 或蛋白质功能分析确认. 还必须考虑可读框内缺失/插入的可能性, 其长度变化较小(PM4), 可以保留蛋白质的关键结构域, 因此导致轻微或中性效应,或功能获得效应.
- 基因会有不同的转录本, 哪一种转录本与生物学功能相关, 在哪些组织会表达哪些转录本, 这些都是需要进行重点考虑的. 如果一个截短变异只限于一个或并非所有转录本, 则必须谨慎考虑到可能存在其他同功型蛋白质, 防止过度解释.
- 如果发现一个无功能变异位于某个外显子上, 而该外显子先前无致病变异报道, 那么该外显子可能被选择性剪切了, 此时需要谨慎考虑该变异的致病性. 当预测的截短变异是偶然发现时(与检测指征无关)则应特别小心, 在这种情况下该位点致病的可能性非常低.
PS1 极强致病性变异
多数情况下, 尤其是当致病机制是蛋白质功能发生改变时, 如已确定某一错义变异是致病变异, 应考虑到与其位于同一变异位点的不同形式的碱基改变也可能产生相同的错义突变结果——氨基酸改变相同(如c.34G>C(p.Val12Leu)和c.34G>T(p. Val12Leu)), 那么, 这些变异也应是致病突变.
需要重点注意:
变异可能不是通过改变氨基酸的水平, 而是通过改变DNA 的序列来发挥作用, 例如, 破坏剪接位点(可通过软件分析确定),
PS2 PM6 新发变异
当将一个新发变异(父母样本检测结果阴性)归类为强的致病证据时, 需要满足以下条件:
- (i) 身份检验表明患者的父母是其生物学父母. 注意如果父母的身份是假定的而没有被证实, 则判定为PM6;
- (ii)患者的家族史符合新发变异特征. 例如, 显性遗传病患者的父母均未患病. 在存在生殖细胞嵌合现象时也可能有1 个以上同胞患病;
- (iii) 患者的表型与变异基因异常引起的表型相吻合.
例如
患者具有特殊面容、多毛和上肢缺陷(即Cornelia de Lange 综合征), 检测到NIPBL 基因的新生突变即为强致病证据, 而患者仅表现为非特异性的发育迟缓, 通过外显子组测序发现的该基因的新发变异, 则判断此变异致病性的证据较弱.
PS3 BS3 功能研究
功能实验研究是一种研究变异致病性的非常强大的工具, 然而并非所有的功能研究都能有效地预测基因或蛋白的功能.
重点注意
功能实验的有效性、重复性和稳定性应重点考虑, 这些参数用来评估功能实验的分析性能以及判定样本诊断信息的完整性, 该完整性容易受标本采集的方法及时间、存储及运输的影响.
评估变异在剪接位点、编码序列、非翻译区以及更深的内含子区域的影响时, 对变异在信使RNA 水平(如信使RNA 的稳定性、加工或翻译)进行评估, 可以提供丰富的信息. 相关的技术方法包括对RNA 和/或互补DNA 衍生物进行直接分析, 以及体外微小基因剪接分析
PS4 PM2 BA1 BS1 BS2 变异频率及对照人群的使用
通过搜索公共人群数据库(如千人基因组数据库, NHLBI 外显子测序数据库, EXAC 数据库; 表1),并利用已发表文献中相同种族的对照数据进行基因变异频率分析(译者注: 此条款在指南更新时会有修改), 通过分析变异基因在对照人群或普通人群中的携带频率, 有助于评估该变异的潜在致病性. NHLBI外显子测序数据库来源于白种人和非裔美国人群,根据其数据覆盖量能够识别是否存在基因变异. 尽管千人基因组数据库缺乏评估基因变异能力, 但它囊括了更多的种族人群, 因此其数据具有更广泛代表性的. EXAC 数据库近期发布了一组来源于不同人群的6 万多个外显子组的等位基因频率数据, 包括了大约三分之二的NHLBI 外显子测序数据. 一般情况下, 某一等位基因在对照人群的频率大于疾病预期人群(表7)时, 可认为是罕见孟德尔疾病良性变异的强证据(BS1), 如果频率超过5%时, 则可认为是良性变异的独立证据(BA1). 此外, 如果疾病发生在早期,且变异在健康成人中以隐性(纯合子)、显性(杂合子)或X-连锁(半合子)的状态存在, 那么这就是良性变异的强证据(BS2). 如果数据库中未能检出变异的存在,应该确认建立该数据库采用的测序读长深度是否足以检测出该位点上的变异. 如果在一个大样本的普通人群或队列数据的对照人群(>1000 人)中变异不存在(或隐性遗传的突变频率是低频), 并且携带此变异的患者与对照人群为同一种族, 那么可以认为该变异是致病性的中等证据(PM2). 许多良性变异是“个体化的”(即个人或家系独有的), 因此即使在相同种族的人群中缺乏也不能作为致病性的充足甚至强的证据
PM1 热点突变和/或关键的、得到确认的功能域
某些蛋白结构域对蛋白质的功能起到了关键作用, 如果在这些结构域上发现的所有错义突变均已被证实为致病突变, 且这些结构域中一定没有已知的良性突变, 那么这就能作为致病的中等证据. 此外, 基因中某些功能尚未确定的区域已被证实存在许多突变热点, 若突变发生在基因突变热点上, 且一个或多个邻近残基中存在较高频率的已知致病突变,那么这也能作为致病的中等证据.
PM3 BP2 顺式/反式检测
检测双亲样本以确定变异在基因上以顺式(incis)(位于基因的同一拷贝)或是反式(in trans)(位于基因的不同拷贝)方式排列, 这对评估变异的致病性非常重要. 例如, 当两个杂合变异发生在隐性遗传病的致病基因上时, 如果已知其中一个变异为致病变异,那么当另一个待分类变异与其呈反式排列时, 这可以作为待分类变异的中等致病证据(PM3). 另外, 若待分类变异与多个已知致病变异均呈反式排列, 则该证据可升级为强致病证据. 但是, 若待分类变异在普通人群中存在, 则需要用统计学方法判断该现象是否为随机共发生事件. 相反, 当已知致病变异与另一个待分类变异呈顺式排列时, 这可以作为待分类变异的良性支持证据(BP2). 如果发生在隐性遗传病致病基因上的两个杂合变异的致病性均未知, 那么确定它们以顺式或是反式排列, 并不能为判断其中任一变异的致病性供更多信息. 但是, 如果两者以顺式排列, 则该基因两个拷贝均受影响的可能性将会降低.对于显性遗传病而言, 若待分类变异与致病变异呈反式排列, 则可作为该变异的良性支持证据(BP2); 对于特定研究成熟的疾病模型, 甚至可以考虑将其作为独立良性证据(如CFTR 相关变异的评估)
PM4 BP3 由于框内缺失/插入和终止密码子丧失导致的蛋白长度改变
相较于单一的错义突变所导致的蛋白质长度变化, 一个或多个氨基酸的缺失或插入、以及由终止密码子变为翻译氨基酸的密码子(如终止密码子丢失)而导致的蛋白质延长更可能破坏蛋白质功能. 因此,框内缺失/插入以及终止密码子丢失可作为中等致病证据. 缺失、插入或延伸范围越大, 缺失区域的氨基酸越保守, 则支持致病的证据越强. 相反, 在重复区域或在进化中不是很保守的区域中小的框内缺失/插入致病的可能性较小
PM5 同一位置新的错义变异
如果一个新发错义突变发生在一已知致病突变导致相同氨基酸改变的位置上( 如Trp38Ser 和Trp38Leu), 那么可作为中等致病证据(但不能假定一定是致病的), 尤其当新的突变比已知致病错义突变更保守时. 此外, 不同的氨基酸变化可能导致不同的表型. 例如, FGFR3 基因编码的Lys650 残基的不同变化与不同的临床表型相关: p.Lys650Gln 或p.Lys650Asn 会导致轻度软骨发育不良; p.Lys650Met会导致严重的软骨发育不全伴发育迟缓和黑棘皮病;p.Lys650Glu 会导致2 型发育异常及致命的骨骼发育不良.
PP1 BS4 共分离分析
在使用家系中变异的共分离现象作为致病性证据时需谨慎. 事实上, 一个与某种表型相关的特定变异在某一家系中的共分离现象是位点与疾病连锁的证据, 而不是变异本身致病性的证据. 一个已经发表的统计方法显示, 在某个家系中鉴定的变异可能与真正的致病变异是连锁不平衡的. 统计模型考虑到了年龄相关的外显率和拟表型率, 一些新的方法也将生物信息分析预测以及与已知致病突变共存作为致病性的单独定量指标. 将远亲纳入统计之中是很重要的, 因为与核心家系成员相比, 他们不太可能同时有该疾病和变异. 对整个基因进行测序(包括整个内含子和5′和3′非编码区)可排查其他致病变异或另一个可能致病的变异的存在. 除非仔细评估基因位点, 否则非致病变异可能被错误地认为是致病变异.当目标基因的特定变异在多个患病的家系成员中以及不同种族背景的多个家系中与表型或疾病共分离时, 则其作为致病的证据不太会受到连锁不平衡和确认偏倚的影响. 在这种情况下, 该标准可以作为中等或强致病证据而不是支持性证据, 其强度取决于共分离的程å度.
另一方面, 一个变异与表型并不共分离时, 为其非致病的强证据. 需要进行仔细的临床评估来排除正常个体的轻度症状和可能的拟表型(患者表型由非遗传或不同的遗传原因引起). 此外, 需确认生物学家庭关系来排除收养、非生父、精子和卵子捐献以及其他非生物学关系. 同时, 外显率下降和年龄依赖性的外显率也必须考虑, 以确保无症状家系成员是真正的无症状.在临床实验室进行共分离的统计评估可能并不容易, 当鉴定了合适的家系时, 为了确保建模合适,并避免得出变异与疾病相关性的错误结论, 鼓励临床实验室与统计或群体遗传学专家合作.
PP2 BP1 变异谱
许多基因具有明确的致病变异和良性变异谱.在某些基因中, 错义突变是导致疾病的常见原因, 且该基因上的良性突变非常少, 那么这种基因上的新发错义突变可作为致病变异的支持证据(PP2). 相反,有些基因致病的唯一已知变异是截短突变, 该基因上的新发错义突变可作为良性的支持证据(BP1). 例如, ASPM 基因的截短变异是该基因引起常染色体隐性遗传小头畸形的主要致病变异类型, 且该基因发生错义多态性突变的频率高, 因此ASPM 基因上的错义变异可认为是良性影响的支持证据.
PP3 BP4 生物信息分析数据
不能过分相信生物信息分析所得到的结果, 特别是不同的生物信息算法依赖于相同或相近的数据进行预测, 并且大多数生物信息算法未被已知致病变异验证过. 此外, 相同算法对不同的基因的预测结果可能完全不同. 如果不同种类算法的分析预测结果一致, 那么生物信息分析结果可以作为支持的证据. 如果绝大多数算法的预测结果不一致, 则这些预测的结果不能用于对变异进行分类. 若某一变异引起的氨基酸改变, 在多个非人哺乳动物物种不太保守的区域中出现, 说明该变异可能不会损害功能, 可以作为良性解读的强的证据. 然而, 如果某基因已在人类中发生进化(如参与免疫功能的基因), 那么在判定该基因在非保守区域中发生的变异为良性时必须小心.
PP4 表型支持
考虑到几乎所有接受疾病针对性测试的患者都有某种表型, 通常, 不将患者表型与某个基因临床特征谱匹配作为判断致病的证据. 但是, 如果满足以下条件, 患者的表型可作为支持证据: (i) 临床检测的灵敏度高, 大多数带有该基因致病突变的患者都被检测为阳性; (ii) 患者有某种明确的综合征的症状,与其他临床表现几乎无重叠(如戈尔林综合征包括基底细胞癌、掌跖坑和牙源性角化); (iii) 该基因通常不存在太多的良性变异(可通过外显子组等人群测序确定的良性变异); (iv) 家族史与疾病遗传方式一致.
PP5 BP6 可靠的来源
现在有越来越多可靠来源(如长期专注于某一疾病领域的临床实验室)的致病性分类信息被分享在数据库中, 但分类判断所依据的证据往往并未供或者很难获取. 在这种情况下, 如果分类信息是近期交的, 那它就可以作为一个单独的支持证据. 然而,还是鼓励实验室共享分类的判断依据, 并与交者进行沟通以评估和创建分类证据. 如果能获得证据,则不应使用这一条款, 而是应该使用相关的证据.
BP5 对共发变异的观察
一般情况下, 当某一变异是在一个有明确的遗传病因的疾病患者中被观察到时, 可作为将该变异解读为良性的证据. 不过, 也有例外. 某一个体可以是某一不相关隐性遗传疾病致病变异的携带者, 因此本证据与隐性遗传性疾病相比, 更支持显性遗传性疾病基因良性变异的分类. 此外, 有些疾病当具有多个变异可以导致更严重的疾病. 例如, 在一个具有严重表型的显性遗传患者中鉴定了两个变异, 一个是致病的, 一个是新的变异, 父母中的一个也有轻微的疾病, 这种情况下, 必须考虑新的变异致病的可能性, 且新的变异使先证者表型加重. 在这种临床情况下, 观察到的第二个新的变异不应分类为良性变异,(尽管在无进一步证据的前下也不认为该变异是致病的). 最后, 有些疾病已知为多基因遗传模式, 如Bardet-Beidel 综合征, 在第二个基因座位上的额外变异也有可能是致病的, 但应谨慎进行报告.
BP7 同义变异
人们逐渐认识到经典的剪接序列以外的剪接错误是一类重要的致病机制, 特别是对那些功能丧失为其常见致病机制的基因. 因此, 在假设同义核苷酸改变没有影响时应持谨慎态度. 然而如果核苷酸位置进化不保守, 且剪接评估算法预测其对剪接一致序列没有影响, 也不会产生新的经典剪接序列, 那么剪接影响的可能性就比较小. 因此, 如果生物信息分析证据支持(BP4), 可将新发同义变异分类为可能良性. 然而, 如果生物信息分析证据表明剪接可能有影响或怀疑有影响(例如, 发生在隐性遗传病致病基因上, 且与已知致病突变呈反式排列的变异), 那么在有功能评估可以供更确切的对影响的评估, 或者得到其他可排除该变异致病作用的证据之前, 该类变异应该归类为意义不明确.

序列变异报告
报告应该使用清晰的语言书写, 避免使用医学遗传学术语, 当必须要使用时需指明所用术语的定义。
报告应包含所有的检测基本要素:,
- 结构化的结果
- 解释
- 参考文献
- 检测方法
- 适当的免责声明.
结果
应根据HGVS 命名规则(见命名部分)列出变异.
基本内容包括:
- 核苷酸(基因组和cDNA)和蛋白质水平的命名
- 基因名称
- 疾病
- 遗传模式
- 外显子
- 合子性
- 变异的分类.
- 若亲本来源明确, 也可包括在内.
当报告外显子组或全基因组测序结果, 或偶尔报告包含基因数目较多的疾病基因包检测结果时, 将变异按“与表型明确相关的疾病基因的变异”、“与表型可能相关的疾病基因的变异”及(在适当情况下)“附带(次要)发现”进行分类可能有益。
解读
解读应包含对变异检测结果进行分类的证据,包括编码蛋白的功能影响预测, 以及检测所发现的变异是否可能全部或部分地解释患者的临床表型.报告也应包括对临床医生的建议, 这些建议包括一些需补充的临床检测, 如对患者进行细胞酶学/功能的检测, 以及对患者家系其他成员进行的变异检测,以便为进一步解读变异检测结果供支持. 解读应当包括检测结果部分述的全部变异, 以及其他附加信息. 对于各个变异需要注明是否已经在先前的文献、疾病病例或对照数据库中有过报道. 在报告结尾处需要列出对变异检测结果分类时所引用的全部参考文献和信息.
方法学
报告中应说明使用的实验方法、检测所涉及的变异类型、检测过程的难点, 以及检测变异所使用的方法的局限性. 需要说明的实验方法应包括核酸的获取方法(如聚合酶链式反应、捕获、全基因组扩增等)以及核酸的检测方法(如双向Sanger 测序、下一代测序、染色体基因芯片、基因分型技术等), 这些信息可以为医务工作者供必要的信息, 以帮助其决定是否需要追加实验来跟进这些检测结果. 方法部分还应包括人类基因组组织基因命名委员会批准的正式基因名称、转录本的RefSeq 登录号和所参考的基因组版本. 对于大的基因包, 基因水平的信息可以通过引用URL 来加以说明. 实验室还可以选择增加对检测过程中常见问题(如样本质量问题、样品混合污染等)的免责声明.
患者维权团体、临床实验和研究的获取
尽管不提倡在实验室报告中对患者供具体临床指导, 但是在报告中供对于检测结果分类的总体信息(如全部阳性检测结果)是恰当且有益的. 大量病人群体和临床试验现在可用于多种疾病的支持和治疗. 实验室可以选择将此信息添加到报告的正文或附加信息, 并且与报告一起发送给医务工作者. 在遵守医疗保险便携性和责任法案(HIPAA)保护患者隐私的前下, 当某一变异检测结果被归为意义不明确时, 实验室可尝试帮助医务工作者和特定的疾病研究小组建立联系.
变异再分析
随着新的变异证据增加, 现有的分类标准需要修订. 例如, 当大样本的有效的人群变异频率被报道后, 许多原本意义不明确的变异, 可以因为明确意义而进行重新分类, 而检测家系中其他成员的结果也可以导致重新分类.
随着检测变异数量的增加及检测范围的扩大,无论是全外显子检测还是全基因组测序, 都可以得到数以百万的变异信息量. 如果实验室缺乏有效的分析方法和足够的文献数据库支撑, 将无法进行变异再分析. 为了满足医务人员和患者的实际需求, 实验室应该开展基因检测数据再分析, 并明确再分析是否产生额外费用. 应该鼓励实验室为帮助医务人员和患者而不断开发更新信息的新途径[31,32].当报告中有针对主要指征的基因中存在临床意义不明的变异, 在实验室又无法及时供更新的数据时, 建议医务人员定期查询其不明意义的变异结果是否被更改. 另一方面, 鼓励实验室在对变异的分类有重要变化时(如致病性或良性的变异被修改)必须主动及时地更新报告. 关于医生对病人报告更新方面的责任, 可详见ACMG 有关指南.
验证
对于孟德尔疾病的致病或可能致病变异需进行正交法验证. 具体方法包括但不限于以下几种: 重新取样和检测、检测父母的变异情况、限制性内切酶消化、对于目标区域重新测序或使用另一种基因分型技术。