本文件适用的体细胞变异类型为单核苷酸变异(SNV)和短片段的插入/缺失变异(Indels),不适用于胚系变异,不适用于融合、拷贝数变异(除了限制在单一基因内的缺失)或其他染色体重排。
本文件适用于对肿瘤基因体细胞序列变异致癌性的判断,是指导肿瘤精准用药的重要环节。肿瘤精准用药全流程包括以下5个步骤:
- 体细胞突变鉴定:
体细胞突变鉴定识别癌症患者个体基因组在体细胞层面发生的变异,如单核苷酸变异(SNV)和短片段的插入/缺失变异(Indels)等。 - 突变注释:
包括确定突变的类型、 位置以及可能影响的基因或蛋白质等,以及通过比对公共数据库,如 ClinVar、 dbSNP 或 HGMD,以了解突变的已知特性和相关研究。 - 突变评级:
突变评级遵循 ACMG/AMP 等相关指南,将突变分为五类:致病性(P)、可能致病性(LP)、意义不明(VUS)、可能良性( LB)和良性(B)。评级基于多条证据,包括
突变的健康人群频率、蛋白功能改变等。 - 临床意义:
评估体细胞突变对患者疾病的具体影响,包括评估突变与疾病的相关性,与特定药物的敏感性或耐药性等。 - 临床决策:
临床医生综合以上信息做出选择用药方案、监测疾病进展、预防性干预或遗传咨询等决策行为。
本文件目标在于指导检测机构对肿瘤体细胞突变的评级,规范体细胞评级中公共数据库或内部数据库的使用,提高肿瘤体细胞评级和精准用药的准确性及有效性性。
证据等级
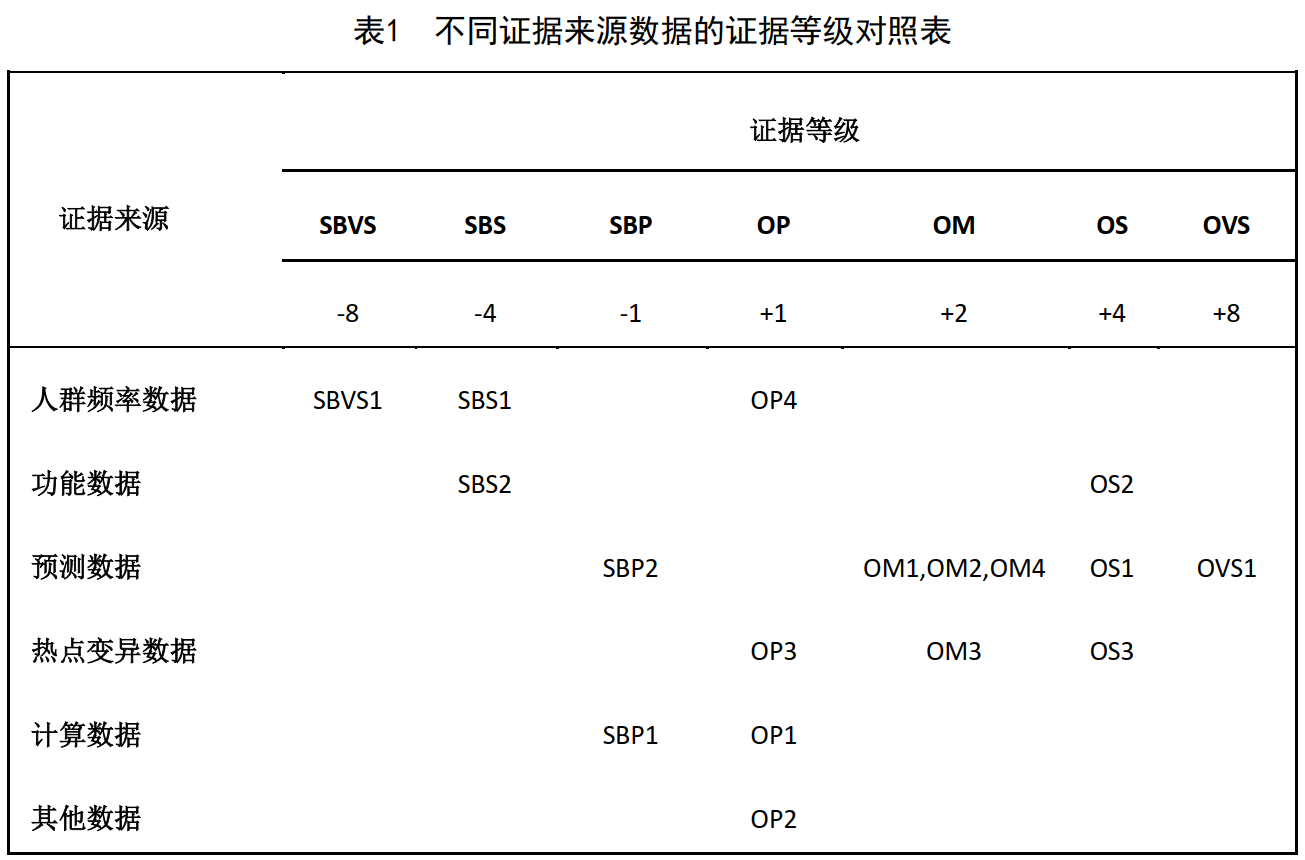
证据项
致癌证据项
人群频率数据
OP4: gnomAD[3] 数据库中正常对照人群中未发现的变异(或频率极低)。
功能数据
OS2: 充分验证的体外或体内的功能研究支持该变异具有致癌效应。注1: 功能实验需要具有重复性与稳定性;注2: 若OS1适用,则只有在功能研究基于特定核苷酸变化时才能使用该证据。
预测证据
OVS1: 抑癌基因发生功能失活的变异, 如无义变异、移码变异、经典±1或±2剪接突变,起始密码子变异等。注1: 具体操作可参考ClinGen工作组提出的PVS1决策树,来对变异的致癌性强弱进行升降级调整[5]。
OS1: 具有与先前已确认的致癌变异相同的氨基酸变化,无论核苷酸的改变如何。需注意剪接影响的改变。
OM1: 位于关键和/或被充分认可的基因功能域,如酶的活性位点。
OM2: 已知的癌基因或抑癌基因中发生编码框内的缺失/插入或在抑癌基因中发生终止密码子丧失,引起的蛋白质长度变化。
OM4: 新的错义变异导致氨基酸变化,同一位置,另一种氨基酸变化已确认是致癌的,并且新的变异在氨基酸改变方面应该与先前被确认为致癌的变异相当或更为显著。注2: 建议采用广受认可的氨基酸差异度量标准,如Grantham's距离、 Epstein's差异系数或Miyata's距离
热点变异证据
OS3: 变异位于热点区域,相同氨基酸位置上的体细胞变异在至少50个样本中观察到,并且相同氨基酸变化的样本数大于等于10。
OM3: 变异位于热点区域,相同氨基酸位置上的体细胞变异在少于50个样本中观察到,并且相同氨基酸变化的样本数大于等于10。
OP3: 变异位于热点区域,并且特定氨基酸变化的样本数少于10个。
计算证据
OP1: 所有使用的生物信息学计算工具均支持变异的致癌作用(保守性/进化性、剪接效应等)。
其他证据
OP2: 为恶性肿瘤已知的单一遗传病因的特定基因的体细胞变异。
良性证据项
人群频率数据
SBVS1: 在人群数据库中任意人群中(如东亚人、非洲人、拉丁美洲人、南亚人、欧洲人(非芬兰人)等),次等位基因的频率≥5%。
SBS1: 在人群数据库中任意人群中(如东亚人、非洲人、拉丁美洲人、南亚人、欧洲人(非芬兰人)等),次等位基因的频率≥1%。
功能数据
SBS2:充分验证的体外或体内功能研究未显示出致癌效应。
预测证据
SBP2: 同义变异,剪切预测算法预测它对剪切保守序列没有影响,也不会产生新的剪切位点,并且该核苷酸并不高度保守。
计算证据
SBP1:所有使用的计算工具均表明该变异对基因或基因产物没有影响(保守性/进化、剪切效应等)。
证据加权规则
各证据强度得分
| 证据强度 | 致癌性 | 良性 |
|---|---|---|
| 非常强 | +8 | -8 |
| 强 | +4 | -4 |
| 中等 | +2 | / |
| 支持 | +1 | -1 |
致癌性分类的得分范围
| 得分范围 | 分类 |
|---|---|
| ≥10 | 致癌 |
| 6-9 | 可能致癌 |
| 0-5 | 意义不明 |
| (-6)-(-1) | 可能良性 |
| ≤-7 | 良性 |
证据不共用
| 证据1 | 证据2 | 不能共用的原因 |
|---|---|---|
| OM1 | OS1 | 都涉及从氨基酸水平评估变异的致癌性 |
| OM1 | OS3 | 都涉及从氨基酸水平评估变异的致癌性 |
| OM3 | OM1 | 都涉及从氨基酸水平评估变异的致癌性 |
| OM3 | OM4 | 都涉及从氨基酸水平评估变异的致癌性 |
| OM4 | OS1 | 都涉及从氨基酸水平评估变异的致癌性 |
| OM4 | OS3 | 都涉及从氨基酸水平评估变异的致癌性 |
| OM4 | OM1 | 都涉及从氨基酸水平评估变异的致癌性 |
| OM2 | OVS1 | 适用的变异类型不同 |
1 | 注1: 如果变异适用OS1证据项,只有功能研究是基于特定核苷酸改变时,才能同时用OS2证据项。 |
知识库
为了提高解读的准确性和解读效率,构建内部知识库已经成为行业内的实际执行标准,该方案也对知识库构建提供了一套宏观的规范和标准。
自行搭建数据库应至少考虑以下几个方面:
- 主要功能或目的(如变异致癌性解读数据库、人群数据库、热点突变数据库、功能学验证数据库登),
- 参考来源,
- 数据采集方式,
- 存储内容与形式,
- 更新维护的方式和频率。
数据库的评价应考虑以下几个方面:
- 完整性(包括1,数据库覆盖的基因范围; 2,数据库是否能覆盖所有证据类型),
- 准确性(确保收录的信息准确无误),
- 时效性(确保收录的信息是最新的)为了提升体细胞变异致癌性解读的准确性,我们引入了多角度的证据类型。不同变异匹配对应的证据需要有一个可靠的知识库支持。
在知识库的搭建过程中,为了不断完善知识库,需要选择合适的变异进行多轮测试,帮助修正知识库中的问题。
结果数据构成
| 序号 | 名称 | 信息内容 |
|---|---|---|
| 1 | 肿瘤类型(Tumor Type) | 肿瘤的命名应符合取得国际公认的国际疾病肿瘤学分类( International Classification of Diseases for Oncology, ICD-O)、国家癌症研究所辞典(National Cancer Institute Thesaurus, NCIt)、统一医学语言系统(Unified Medical Language System, UMLS)或同类型肿瘤学分类标准和命名规则 |
| 2 | 基因(Gene) | 基因应以 HGNC approved symbol 应为标准名 |
| 3 | 变异类型(Mutation Type) | 应明确突变类型为单核苷酸变异(SNV)、短片的段插入/缺失突变(Indels),具体详见附录 A |
| 4 | 变异命名描述(Mutation Description) | 对于单核苷酸变异(SNV)、短片的段插入/缺失突变(Indels),应按照人类基因组协会(Human Genome Variation Society, HGVS)命名规范进行统一的命名,包括碱基改变和氨基酸改变。 |
| 5 | 变异来源(Mutation Origin) | 应明确证据适用胚系突变和/或体细胞突变 |
| 6 | 证据类型(Evidence type) | 分为致癌性证据和良性证据 |
| 7 | 证据强度(Evidence strength) | 分为非常强、强、中等和支持 |
| 8 | 证据等级(Evidence level) | 分为非常强致癌证据(Oncogenic Very Strong, OVS)、强致癌证据(Oncogenic Strong, OS)、中等致癌证据(Oncogenic Moderate, OM)、支持致癌证据(Oncogenic Supporting, OP)、非常强良性证据(Somatic Benign Very Strong, SBVS)、强良性支持证据(Somatic Benign Strong, SBS)和支持良性证据(Somatic Benign Supporting, SBP) |
| 9 | 证据来源(Source of evidence) | 分为人群频率数据、功能数据、预测数据、热点变异数据、计算数据和其他数据 |
| 10 | 致癌性(Oncogenicity) | 分为致癌、可能致癌、意义不明、可能良性、良性 |
| 11 | 提交者(Created user) | 应记录提交人姓名或机构名称 |
| 12 | 提交时间(Created time) | 应记录提交证据记录的日期和时间 |
| 13 | 审核者(Review user) | 应记录审核人姓名或机构名称 |
| 14 | 审核时间(Review time) | 应记录审核证据记录的日期和时间 |
测试数据集
进行这种大范围开发,测试可能是我们最头疼的部分,毕竟如何用最少的数据集合,来最大范围的对我们的体系和方法进行测试验收,是开发阶段更古不变的难题。指南推荐的是ClinGen提供的10各基因的94个突变作为评测标准。
附录
变异类型
本来不想写这部分的,但是有个指标终于算是在一个相对官方的文章中出现了,那就是InDel和CNV到底是怎么界定,之前虽然也一直是按50bp处理(wiki建议)但是总归有些师出无名(或者这个名认可度不够~~),也有一些方案是按30bp处理,类似变异检测软件可能直接整出来上百bp的InDel。本指南对变异类型进行了长度的明确。
| 变异类型 | 变异类型说明 |
|---|---|
| WT | 基因表达正常或基因为野生型 |
| MUT | 涵盖基因下所有变异类型。当证据适用于某基因或某外显子/内含子内全部变异时采用。 |
| SNV+INDEL | 涵盖基因下 SNV、 INDEL 及其子类的所有突变类型 |
| INDEL | 小片段插入或缺失突变(≤50bp) |
| DEL | 小片段缺失突变(≤50bp) |
| DUP | 小片段重复突变(≤50bp) |
| DELINS | 小片段插入缺失突变(≤50bp) |
| INS | 小片段插入突变(≤50bp) |
| SNV | 单个碱基突变 |
| ExonDEL | 基因内一个或多个外显子缺失 |
体细胞评级常用数据库

体细胞评级常用预测软件
