执行标准
某些蛋白结构域对蛋白质的功能起到了关键作用, 如果在这些结构域上发现的所有错义突变均已被证实为致病突变, 且这些结构域中一定没有已知的良性突变, 那么这就能作为致病的中等证据. 此外, 基因中某些功能尚未确定的区域已被证实存在许多突变热点, 若突变发生在基因突变热点上, 且一个或多个邻近残基中存在较高频率的已知致病突变,那么这也能作为致病的中等证据.
按照最新文献报道(PMID: 38645134)使用MPC(错义有害性度量),评价PM1证据项适用性(通过查看gnomAD(gnomAD v2.1.1)中的MCR missense OE参数,当OE≤0.2112时可以使用PM1,当0.2112<OE ≤0.3747时可以使用PM1_Supporting)。
相关基因的oe值可以在 GnomAD 网页获取(注意使用 Regional missense constraint 中提供的每个基因分段的OE值,而非基因水平的oe值)。
具体证据项强度阈值如下: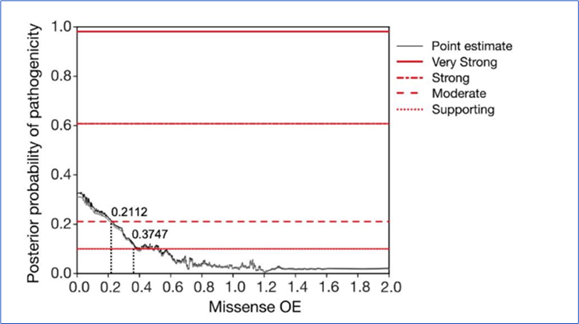
数据获取
下载文件处理
其中OE值可以直接从gnomAD中获取:
- 对基因内的不同区域根据选择压力差异进行分区计算oe值的结果:gnomAD_v2.1.1_transcripts_with_rmc.tsv。
- 基因水平oe值,不对基因进行分段的结果:gnomad.v2.1.1.lof_metrics.by_gene.txt.bgz
1 | gnomad.v2.1.1.lof_metrics.by_gene.txt.bgz 有全部基因的 z-score 值,和对应基因水平的 mis_oe 值(如果分段数据中没有记录的话,直接使用基因水平oe值)。 |
获得上述文件后,通过脚本
api遍历
由于上述信息不全,经过查询,gnomAD本身提供了API查询接口,可以用于信息的获取。
以下为一个python示例
1 | # This example uses the GQL GraphQL client library. |
会得到gnomAD的数据,其中包含我们在query中所列出的相关信息基因的z-score、oe值、oe值区间等。BRCA1 和 HERC2 的查询示例结果如下:1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
30
31
32
33
34
35
36
37// BRCA1
{'gene': {'gene_id': 'ENSG00000012048',
'symbol': 'BRCA1',
'ncbi_id': '672',
'chrom': '17',
'start': 41196312,
'stop': 41277500
'gnomad_constraint': {'oe_mis': 0.9466031983859604,
'oe_mis_lower': 0.895,
'oe_mis_upper': 1,
'mis_z': 0.5821512027320781
},'gnomad_v2_regional_missense_constraint': {'regions': []}}}
// HERC2
{'gene': {'gene_id': 'ENSG00000128731',
'symbol': 'HERC2',
'ncbi_id': '8924',
'chrom': '15',
'start': 28356186,
'stop': 28567298
'gnomad_constraint': {'oe_mis': 0.7629974287956002,
'oe_mis_lower': 0.735,
'oe_mis_upper': 0.791,
'mis_z': 4.423675453049803
},'gnomad_v2_regional_missense_constraint': {'regions': [
{'chrom': '15', 'start': 28356912, 'stop': 28361934, 'obs_exp': 0.6411651984433206},
{'chrom': '15', 'start': 28361935, 'stop': 28362185, 'obs_exp': 0},
{'chrom': '15', 'start': 28362186, 'stop': 28397963, 'obs_exp': 0.5991045884304614},
{'chrom': '15', 'start': 28397964, 'stop': 28414784, 'obs_exp': 0.965845689035514},
{'chrom': '15', 'start': 28414785, 'stop': 28424152, 'obs_exp': 0.36527103011859574},
{'chrom': '15', 'start': 28424153, 'stop': 28441647, 'obs_exp': 0.650862288680692},
{'chrom': '15', 'start': 28441648, 'stop': 28479373, 'obs_exp': 1.028254211028902},
{'chrom': '15', 'start': 28479374, 'stop': 28493711, 'obs_exp': 0.6391324339997898},
{'chrom': '15', 'start': 28493712, 'stop': 28502338, 'obs_exp': 0.9025883099911312},
{'chrom': '15', 'start': 28502339, 'stop': 28514578, 'obs_exp': 0.5778393805973672},
{'chrom': '15', 'start': 28514579, 'stop': 28566579, 'obs_exp': 0.9959412526339723}
]}}}
oe值的计算: 参考文章:The landscape of regional missense mutational intolerance quantified from 125,748 exomes